NetworkX 배우기(with python)
NetworkX
NetworkX는 파이썬 기반의 모듈로, 다양한 그래프 알고리즘을 제공한다.
이를 이용해서 노드와 엣지로 연결된 네트워크를 분석할 수 있다.
우선, 이렇게 networkx를 nx라는 이름으로 가져와서 그래프를 작성할 환경을 준비한다.
import networkx as nx
G = nx.Graph
1. 노드/엣지의 삽입
이제 준비된 빈 그래프에 노드 1개를 만들고 그 내용을 출력해보았다.
노드를 삽입할 때는 add_node() 라는 메소드를 사용하고, 값을 입력하면 해당 노드의 라벨(lable)로 지정된다.
nodes() 메소드를 사용하면 그래프에 포함된 모든 노드를 출력할 수 있다.
G.add_node(1)
print(G.nodes())
출력 결과는 1이다!

문자나 문자열도 라벨로 지정할 수 있다.
G.add_node('P')
G.add_node('Hi')
print(G.nodes())
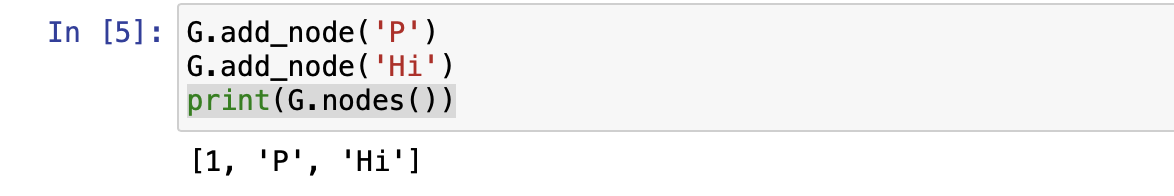 그래프에 포함된 모든 노드를 출력하면 [1, ‘P’, ‘Hi’] 가 나온다.
그래프에 포함된 모든 노드를 출력하면 [1, ‘P’, ‘Hi’] 가 나온다.
그럼 한 번에 여러 개의 노드를 삽입할 수는 없을까?
add_nodes_from() 메소드를 사용하면 리스트 형태의 데이터 입력이 가능하다!
G.add_nodes_from([2, 3])
print(G.nodes())

2, 3 노드가 한 번에 추가되었다.
지금까지 작성된 그래프 G에 입력된 노드들을 한 줄씩 출력해보자.
for node in G.nodes(): #그래프의 모든 노드를 한 줄씩 출력
print(node)

그렇다면, 이미 존재하는 노드를 다시 추가하면 어떻게 될까?
G.add_nodes_from([3,4])
print(G.nodes())

출력 결과를 보면 3의 노드는 이미 존재하기 때문에 4의 노드만 추가된 것을 확인할 수 있다.
2. 그래프를 그림으로 출력하기
그래프를 리스트로만 보려니 답답하다!
그래프를 그림으로 출력해보자.
draw() 메소드를 사용하면 된다.
이 때, with_labels=True는 라벨을 노드 위에 함께 출력한다는 의미이다. False를 넣으면 라벨은 생략하고 출력된다.
nx.draw(G, with_labels = True, node_color='lightblue')

이렇게 둥근 노드 위에 각각의 라벨이 표시되어 출력되었다.
이제 노드와 노드 사이를 연결하는 선인 엣지를 입력해보자.
add_edge() 메소드를 사용한다.
edge_color()을 추가해 엣지의 색상도 지정할 수 있다.
G.add_edge(1,2)
nx.draw(G, with_labels=True, node_color='lightblue', edge_color='grey')

1의 노드와 2의 노드를 연결하는 엣지가 생겼다!
노드의 연결 상태를 라스트 형태로도 확인 가능하다.
print(G.edges()) #연결 상태를 리스트 형태로 확인

만약 엣지를 연결할 때 기존에 없던 노드를 연결하면 어떻게 될까?
G.add_edge(4,5) #존재하지 않는 5의 노드와 연결
print(G.edges()) # 5의 노드 생성 후 4와 연결

위의 코드처럼 기존의 노드를 새로운 노드와 연결하는 엣지를 추가하면, 새로운 노드를 생성한 후 연결한다.
add_nodes_from() 메소드처럼 add_edges_from()메소드를 이용해 여러 개의 엣지들을 한 번에 만들 수 있다.
1개 이상의 튜플을 리스트 형식으로 묶어서 입력하면 된다.
1과 P노드 사이의 엣지와 1과 Hi 노드 사이의 엣지를 만든 후, 전체 그래프를 그리는 코드를 작성해봤다.
G.add_edges_from([(1, 'P'), (1, 'Hi')])
nx.draw(G, with_labels=True, node_color='yellow', edge_color="red")
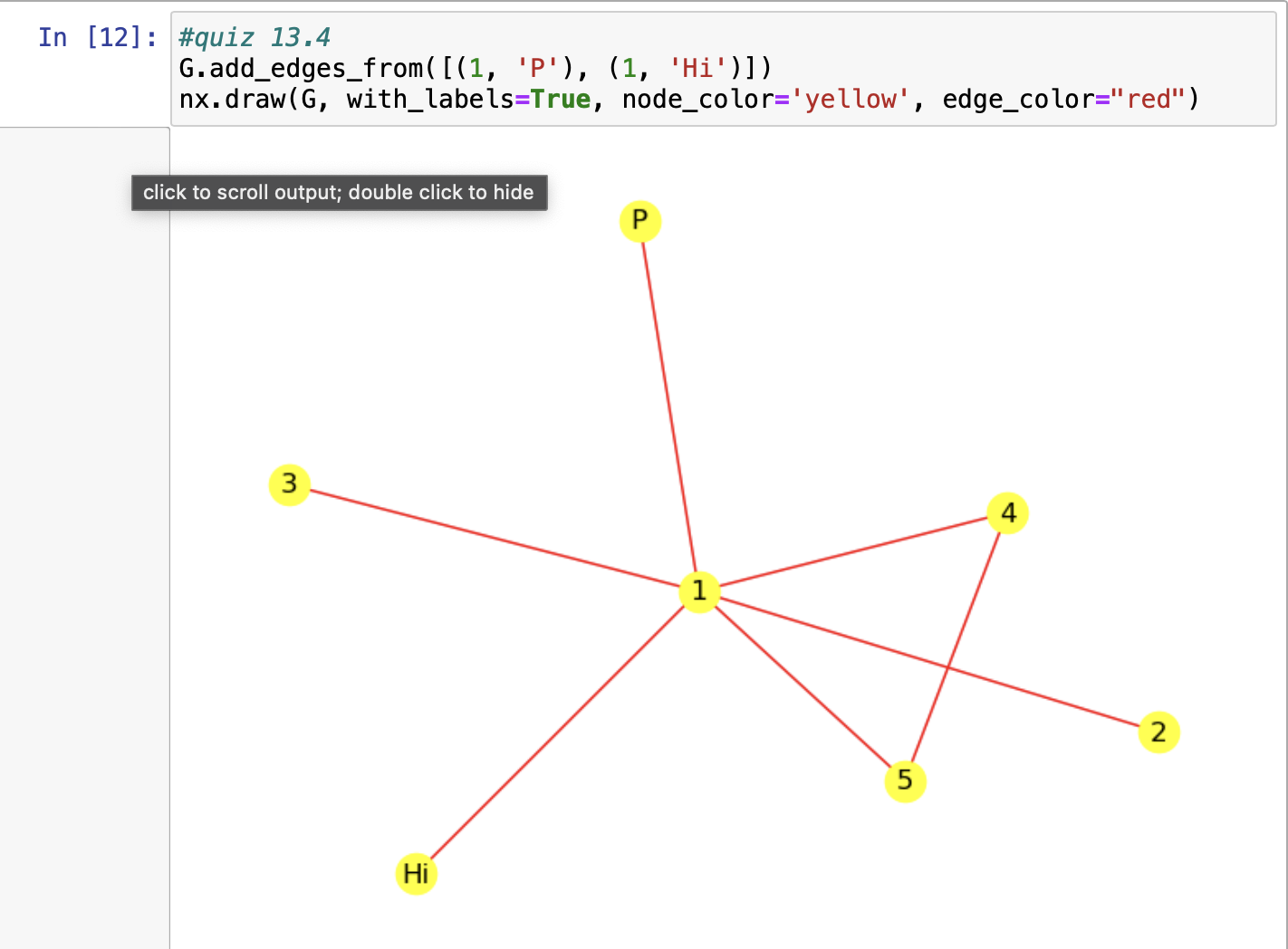
3. 다수의 노드와 엣지를 동시에 생성하기
path_graph() 와 add_path() 메소드를 사용할 수 있다.
path_graph() 메소드
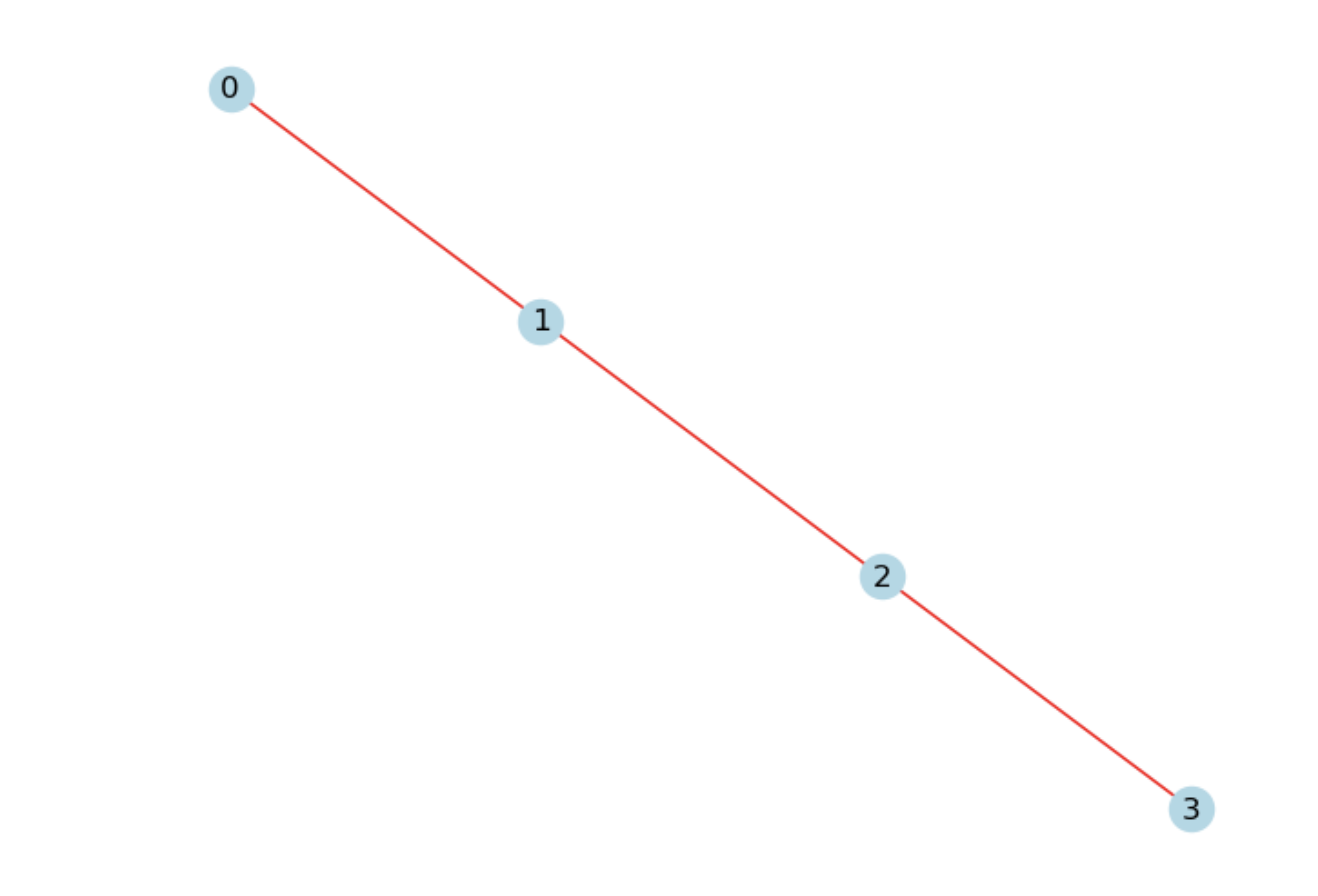
먼저, 아래의 예시처럼 path_graph(4)를 입력하면 0부터 3까지의 노드를 생성하고 그들을 잇는 엣지들이 만들어진다.
import networkx as nx
G = nx.path_graph(4)
nx.draw(G, with_labels=True, node_color='lightblue', edge_color="red" )
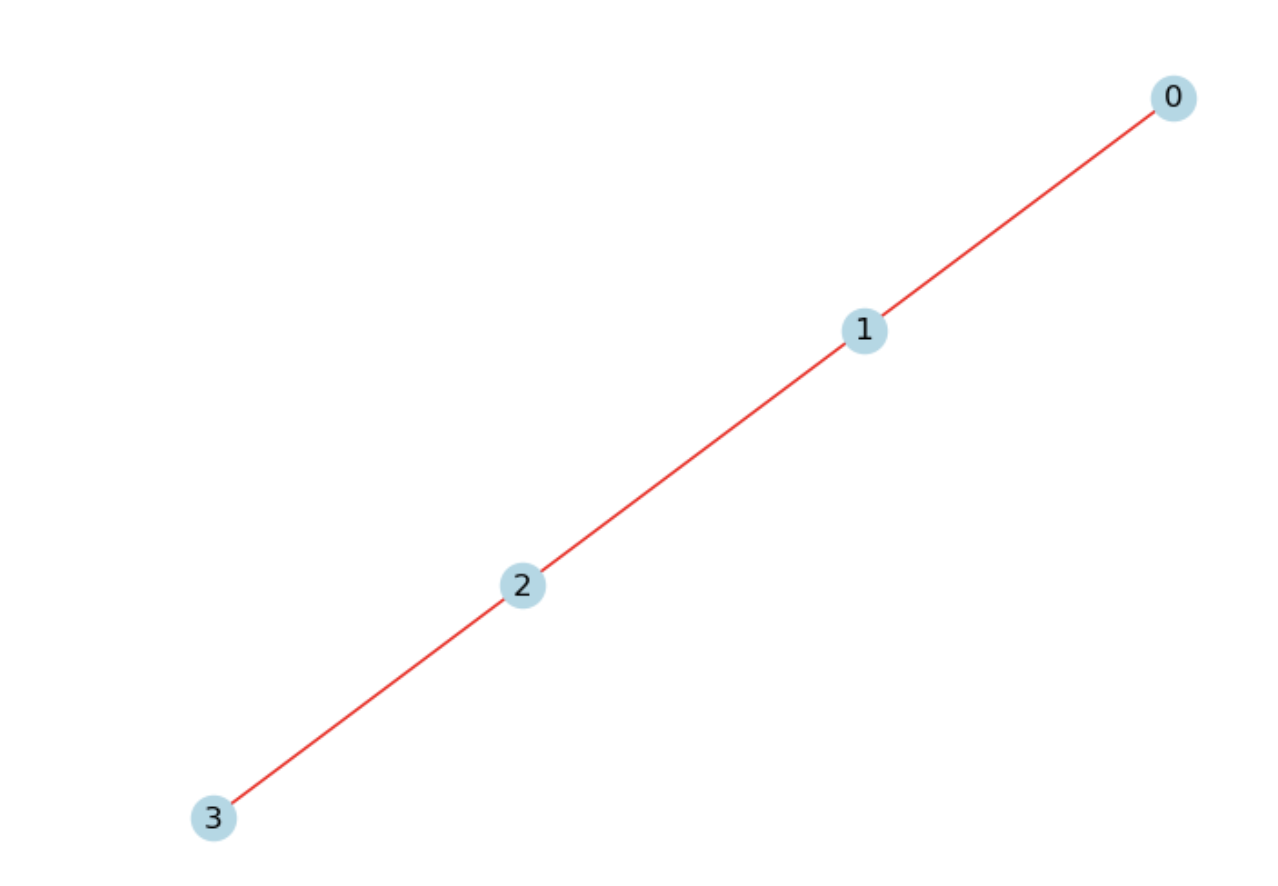
add_path() 메소드
add_path() 메소드에 리스트를 입력해 노드와 엣지를 생성할 수도 있다.
G = nx.Graph()
nx.add_path(G, [0,1,2,3])
nx.draw(G, with_labels=True, node_color='lightblue', edge_color="red" )
이렇게 작성하면 그래프 G에 0,1,2,3의 노드들이 생성되고 이들을 잇는 엣지도 자동으로 생성된다.
4. 노드의 degree(차수) 구하기
degree(차수)는 특정 노드에 연결된 엣지의 개수를 의미한다.
degree()메소드를 사용해 모든 노드의 차수를 구할 수 있는데,
사전 형식으로 모든 노드에 대한 차수를 쌍으로 저장한다.
예시 코드를 통해 자세히 알아보자!
주석을 달아 설명을 덧붙였다.
G = nx.Graph()
nx.add_path(G, [0,1,2,3])
print(G.degree(0)) #노드 0에 대한 차수만 출력
print(G.degree([0,1])) #노드 0,1 에 대한 차수 출력 (1,2)는 노드 1이 엣지 2개를 가져서 degree=2 라는 의미.
print(G.degree()) #모든 노드에 대한 차수 출력
출력 결과

5. 노드 / 엣지의 삭제
필요 없는 노드나 엣지를 삭제해보자.
노드 삭제
remove_node() 메소드를 이용한다.
삭제하고자 하는 노드의 라벨을 입력하면 된다.
예시로 2의 노드를 삭제해보겠다.
print(G.nodes())
G.remove_node((2))
print(G.nodes())
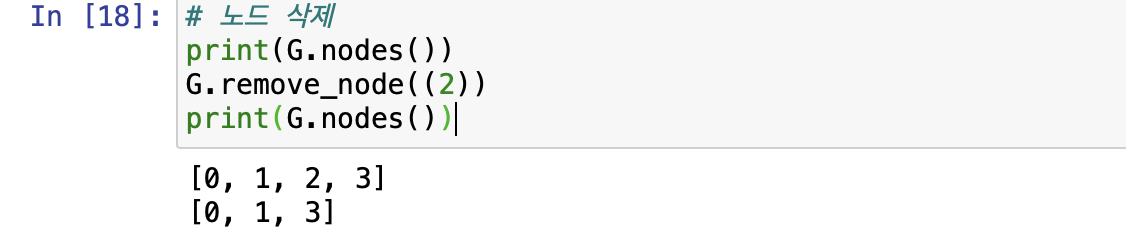
출력 결과를 보면, 2의 노드가 잘 삭제되었다.
다수의 노드를 한꺼번에 삭제할 수는 없을까?
물론… 가능하다.
remove_nodes_from() 메소드를 사용, 리스트 형태로 입력한다.
예시 코드를 보자.
print(G.nodes())
G.remove_nodes_from([0,1,3])
print(G.nodes())

이렇게 한꺼번에 모든 노드를 삭제할 수 있다.
엣지 삭제
엣지를 삭제하는 방법도 노드의 경우와 아주 유사하다.
remove_edge() 혹은 remove_edges_from() 메소드를 사용한다.
노드 삭제와 마찬가지로 전자는 1개의 엣지를 삭제할 때 사용하고, 후자는 여러 개의 엣지를 한 번에 삭제할 때 사용할 수 있다.
print(G.edges())
G.remove_edge(1,2)
print(G.edges())
1 노드와 2 노드 사이의 엣지를 삭제하도록 하고 출력 결과를 보면,

잘 삭제되었다.
다수의 엣지를 한 번에 삭제하는 경우도 살펴보자.
print(G.edges())
G.remove_edges_from([(0,1), (2,3)])
print(G.edges())

5. 노드 / 엣지의 개수
생성한 노드나 엣지들의 개수를 알고싶다면
number_of_nodes()나 number_of_edges() 메소드를 사용한다.
예시 코드를 살펴보자!
import networkx as nx
G = nx.Graph()
G.add_nodes_from([1, 2, 3, 4, 5])
G.add_edges_from([(1,2), (1,3), (1,4), (1,5), (4,5)])
print('No. nodes:', G.number_of_nodes())
print('No. edges:', G.number_of_edges())

노드와 엣지의 개수가 각각 5개로 잘 출력된다.
그런데, 노드를 삭제하면 연결된 엣지도 어떻게 될까?
노드의 생성과 삭제 과정을 거쳐 노드와 엣지의 개수를 비교해보았다.
import networkx as nx
G = nx.Graph()
G.add_nodes_from([1, 2, 3, 4, 5])
G.add_edges_from([(1,2), (1,3), (1,4), (1,5), (4,5)])
G.remove_node(3) # 노드를 삭제하면 3 노드와 연결되어있던 엣지도 자동으로 삭제됨
print('No. nodes:', G.number_of_nodes())
print('No. edges:', G.number_of_edges())

결과를 보면, 삭제된 3의 노드에 연결되어 있던 1-3 엣지도 삭제되었음을 알 수 있다!
6. 특정 확률분포의 그래프 생성하기
networkx 를 통해 다양한 확률분포 모델의 그래프를 생성할 수 있다!
erdos_renyi_graph(), watts_strogatz_graph(), barabasi_albert_graph() 등이 있다.
각각의 메소드는 입력 파라미터를 가지고 있고, N(노드 개수)과 p(노드 간 연결확률)을 입력한다.
이 중 가장 간단한 알고리즘인 erdos_renyi_graph()를 이용해 그래프를 출력해보자.
import networkx as nx
import matplotlib.pyplot as plt
G = nx.erdos_renyi_graph(25, 0.2)
nx.draw(G)
25개의 노드가 생성되고, 노드 간의 엣지가 생길 확률은 0.2이다.
이런 그래프가 출력된다.
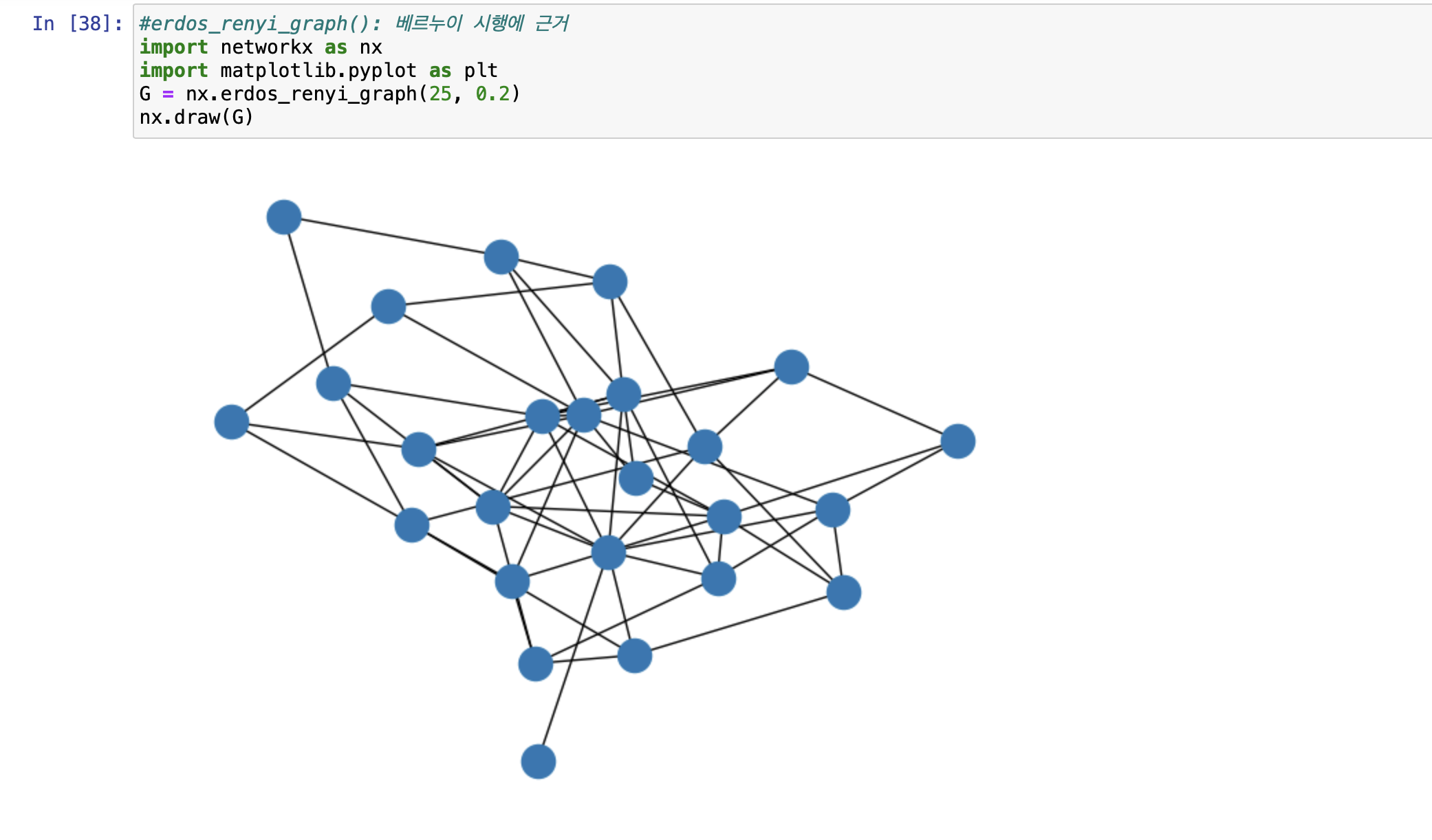
7. erdos_renyi_graph()
erdos_renyi_graph()에 대해 좀 더 자세히 알아보자.
이는 베르누이 시행에 근거한 방식이다.
예를 들면, 일정한 확률을 주고 동전의 앞면이나 뒷면이 나오게 하는 방법이다.
베르누의의 기능은 scipy.stats에서 제공해주고, rvs() 메소드를 사용하면 특정 확률을 입력해 결과를 얻을 수 있다.
그럼 동전을 6번 던져서 나오는 결과들을 확인해보자!
확률은 0.33으로 설정했기 때문에 6번의 시행 중 1이 나올 확률은 2번 정도이다.
from scipy.stats import bernoulli
for i in range(6):
print(bernoulli.rvs(p=0.33))
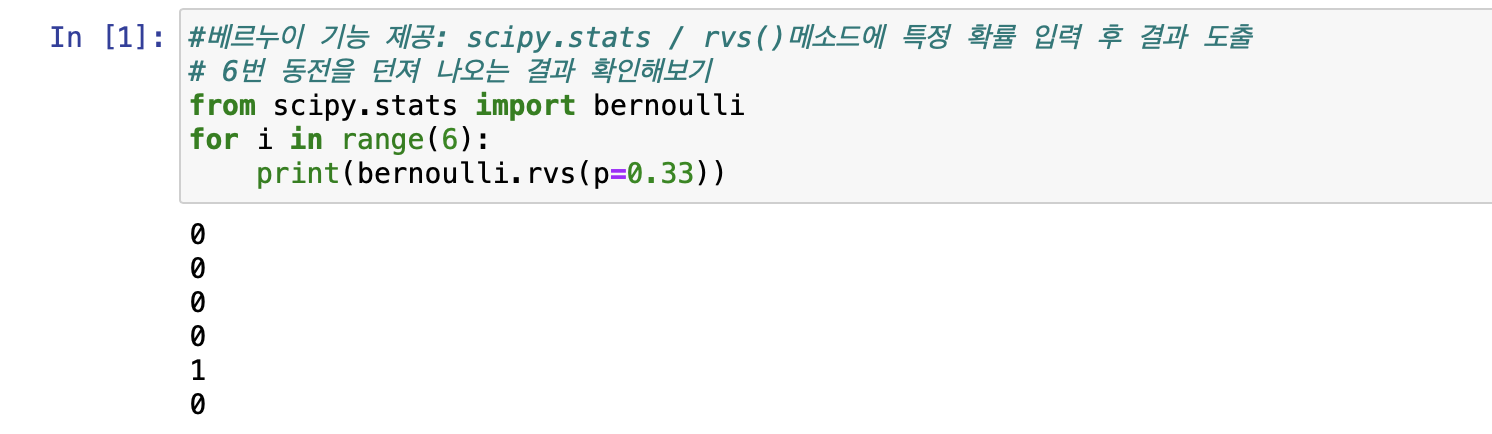
이번엔 1이 한 번 나왔다!
이제 erdos_renyi_graph() 메소드 기능을 하는 함수를 만들어보자.
이번에도 주석을 달아 자세한 설명을 덧붙였다.
import numpy as np
import networkx as nx
import matplotlib.pyplot as plt
from scipy.stats import bernoulli
def erdosGraph(N, p):
G = nx.Graph()
G.add_nodes_from(range(N))
listG = list(G.nodes())
for i, node1 in enumerate(listG): # 모든 노드 쌍에 대해 반복
for node2 in range(node1 + 1, N): # 중복을 피하기 위해 node1 이후의 노드만 고려
if bernoulli.rvs(p=p): # 확률 p로 두 노드를 연결할지 결정
G.add_edge(node1, node2) # 노드를 연결
return G
nx.draw(erdosGraph(20, 0.18))
이런 코드를 작성하면 20개의 노드에서 0.18의 확률로 노드 간 엣지가 생성된 그래프가 출력된다.
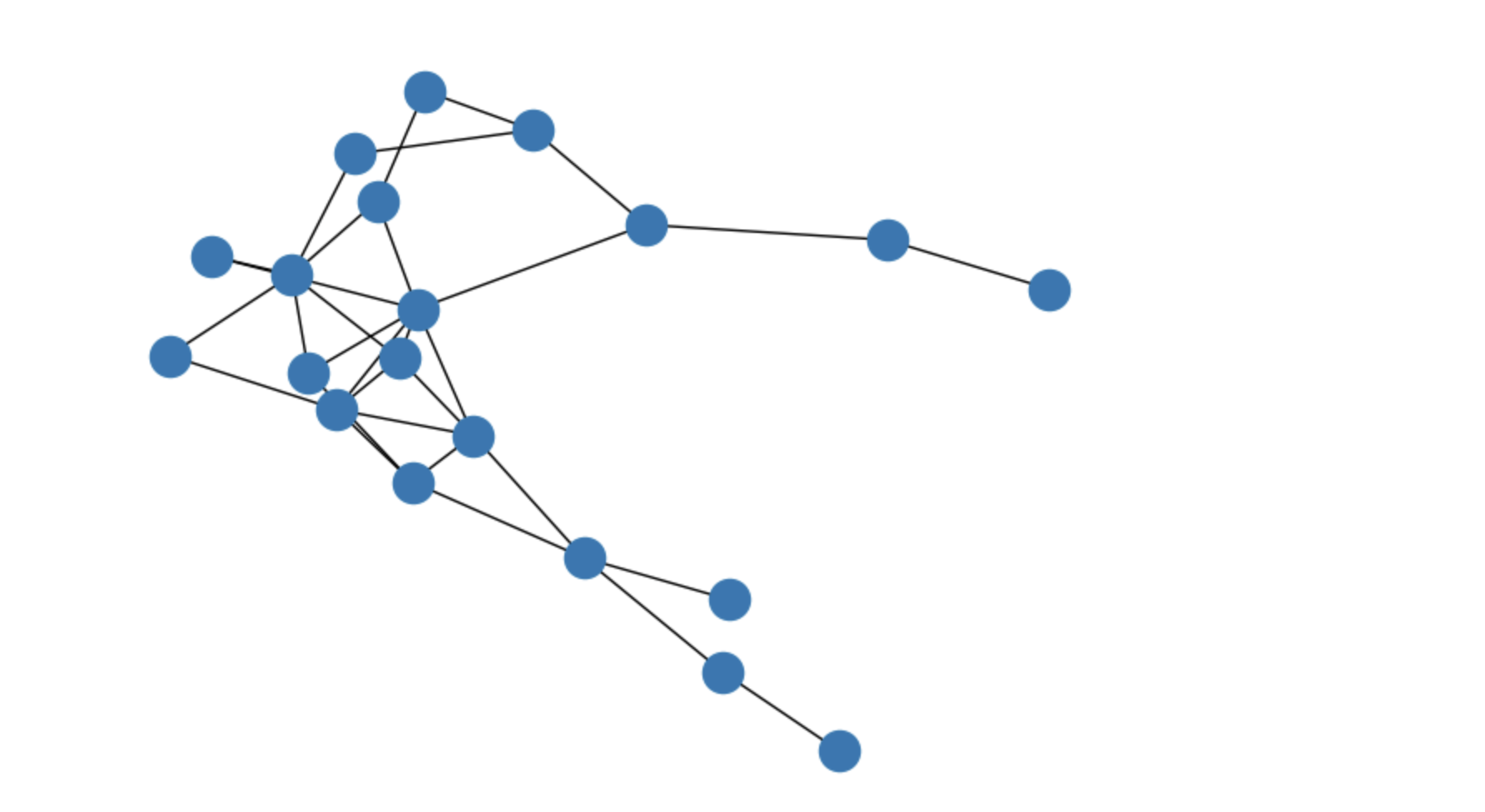
이 부분에서 enumerate()함수의 개념이 조금 헷갈려서 찾아봤다.
간단히 말하면, enumerate()함수는 인덱스와 원소로 이루어진 튜플을 만들어준다.
각각의 원소에 인덱스를 부여해 저장해주는 것이다.
참고 이에 대해 자세하게 설명해둔 페이지가 있어 링크를 첨부한다.
👉 https://www.daleseo.com/python-enumerate
루프와 enumerate()함수의 원리에 대해서도 알아볼 수 있다.
지금까지의 개념들을 이용해 확률적인 히스토그램을 그릴 수도 있다.
역시 erdos_renyi_graph()를 이용해 코드를 작성해보겠다.
import numpy as np
import networkx as nx
import matplotlib.pyplot as plt
from scipy.stats import bernoulli
def erdosGraph(N, p):
G = nx.Graph()
G.add_nodes_from(range(N))
listG = list(G.nodes())
for i, node1 in enumerate(listG): # 모든 노드 쌍에 대해 반복
for node2 in listG[i+1:]: # 중복을 피하기 위해 node1 이후의 노드만 고려
if bernoulli.rvs(p=p): # 확률 p로 두 노드를 연결할지 결정
G.add_edge(node1, node2) # 노드를 연결
return G
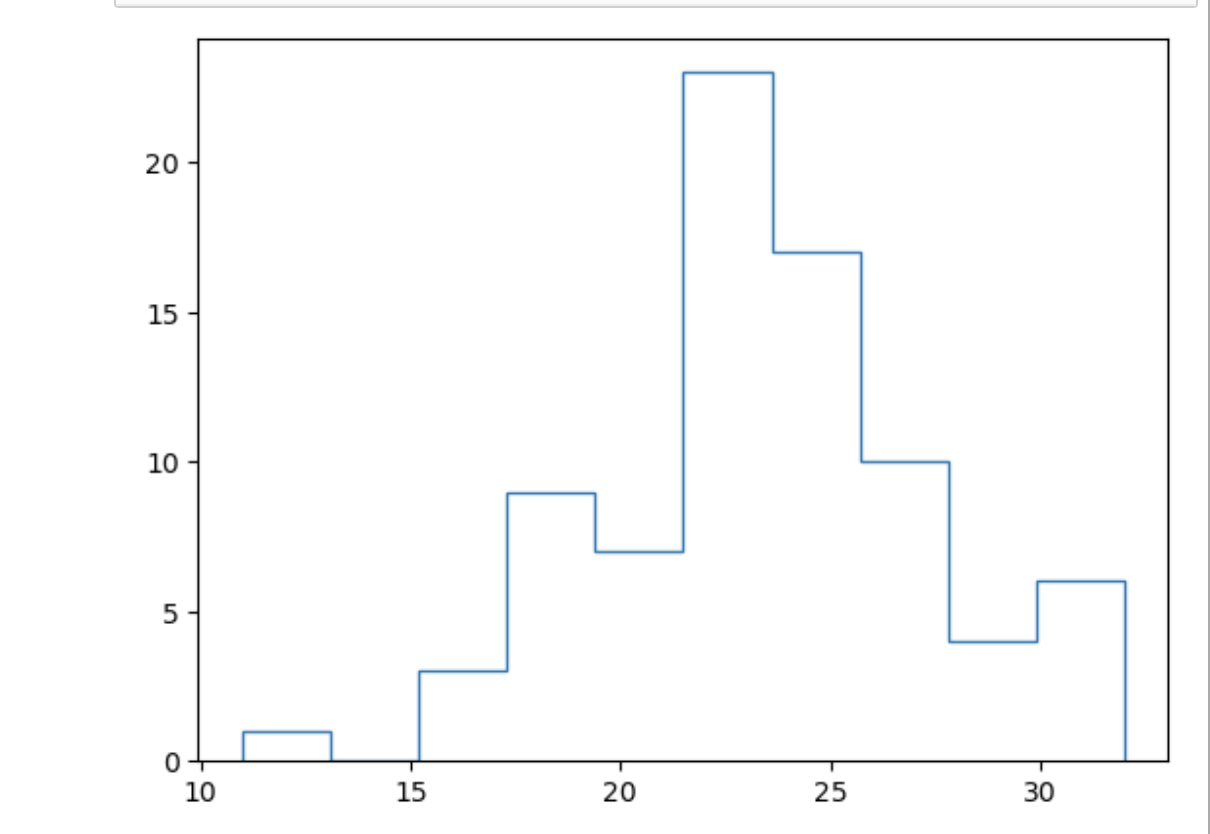
G1 = erdosGraph(80, 0.3)
plt.hist(list([d for n, d in G1.degree()]), histtype='step'); #히스토그램 그리기
8. 엑셀 파일로 그래프 생성
엑셀 파일에 작성된 행렬로 그래프를 생성해보자.
먼저, x*y 행렬에서 인덱스 번호인 i와 j값이 같은 위치의 값들은 모두 동일한 값으로 배치해야 한다.
무슨 말인지 잘 와닫지 않을 수도 있다.
예시를 들어보겠다.
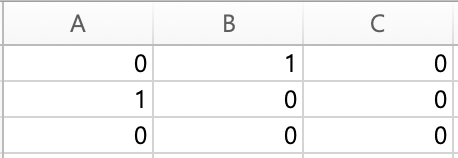
각 셀에 저장된 값은 0 혹은 1이다.
(0,1)의 값이 1이라면 0의 노드와 1의 노드가 연결되어 있다는 의미이다.
이 때, 0과 1이 연결되어 있다면 당연히 1과 0도 연결되어있으므로
(1.0)의 값도 1이어야 한다 !!
이제 엑셀에 이런 값들을 입력하고 CSV 파일로 저장하자. (나는 “test1(networkx).csv” 라는 이름으로 저장했다.)
이 때, 현재 작업 중인 폴더에 저장해줘야 한다.
그 후 엑셀 파일을 불러와 그래프를 출력해본다.
import numpy as np
import networkx as nx
k = np.loadtxt("test1(networkx).csv", delimiter=",")
G = nx.to_networkx_graph(k)
nx.draw(G, with_labels=True, node_color='yellow', edge_color='red')

출력 결과를 보면 행렬이 3 x 3 이므로 0, 1, 2 세 개의 노드가 생성되었다.
그리고 (0,1)과 (1,0)의 위치의 값이 1이므로 노드 0과 1이 연결되었고
2는 연결된 엣지가 없는 상태로 출력됐다.
지금까지 networkx()의 기본적인 기능들을 소개해보았다.
다음 포스팅에서는 위의 기능들을 활용해 샘플 데이터를 분석해보겠다!
